导读
遗传密码扩展,是通过将非经典氨基酸(ncAAs)引入中心法则,为研究和操控生物过程提供基础工具。
已经证明,遗传密码扩展需要两个组成部分:A)异源转tRNA-合成酶对;B)经过工程化处理可接受研究者定义的ncAAs,以及在翻译过程中可供分配给ncAAs的“空白”密码子。
以往选择的密码子是UAG终止密码子,并且大量研究工作集中在通过菌株和核糖体工程来改善依赖UAG的ncAA掺入。然而,未分配密码子的可用性一直是瓶颈,近期的基因组合成和人工核碱基技术部分解决了这一问题,但其复杂性较高。
尽管许多翻译调控层面(例如密码子序列、tRNA氨酰化和核糖体成分)已被研究来解决这些限制并改善ncAA掺入效果,但对mRNA的研究相对较少。
2024年9月11日,美国斯克里普斯研究所Ahmed H. Badran团队在Nature Biotechnology上发表了一篇题为“Efficient genetic code expansion without host genome modifications”的论文,探讨了mRNA密码子使用对四联解码的影响,发现四联密码子下游紧邻高使用率三联密码子有助于提高靶向解码,并且实现了在不改变宿主细胞的情况下高效引入多种ncAAs,并成功用于体内肽环合成,推动了体外与体内遗传密码扩展的融合。

文章索引
【标题】Efficient genetic code expansion without host genome modifications
【发表期刊】Nature Biotechnology
【发表日期】2024年9月11日
【作者及团队】美国斯克里普斯研究所Ahmed H. Badran团队
【IF】46.9
研究结果
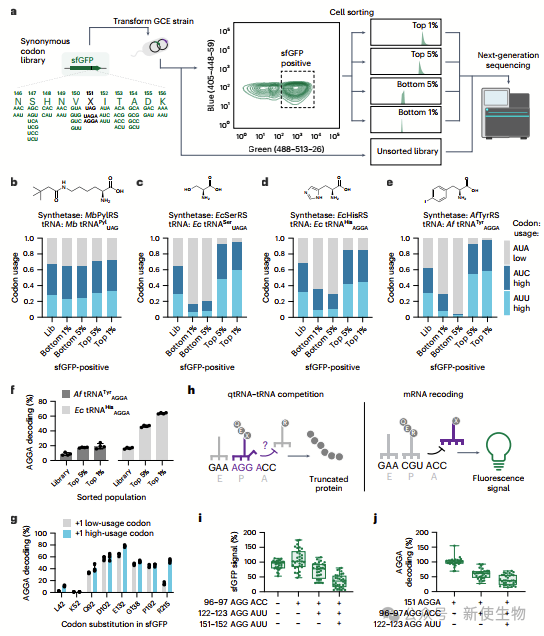
一. 近端和远端密码子使用影响四联密码子解码
通过研究sfGFP报告基因,作者发现四联密码子解码对上下游密码子的使用更为敏感,尤其是下游的+1位置,偏好高频密码子。
为了减少错误解码,他们将报告基因重新编码为高使用率密码子,结果显示解码效率提高且蛋白质纯度增加,表明密码子使用对四联密码子解码至关重要。
二. tRNA-合成酶对在重新编码的通路中表现出活性
作者合成并测试了PylRS–tRNAPylCUA,发现其活性与优化后的大肠杆菌相当。通过测试25种合成酶和32种tRNAs(800种组合),识别出69对高效的tRNA-合成酶对,分为12个互不干扰的集群,确保了密码子兼容ncAA和标准氨基酸的掺入。

三. qtRNA的定向进化可以改善四联密码子解码
为实现多重ncAA掺入,作者优化了5对tRNA-合成酶对的四联密码子解码效率。通过定向进化和系统筛选,作者将解码效率提升至最高100%,并开发了新的四联密码子CGGA。最终,这些优化后的qtRNA-合成酶对能有效地在活细胞中实现ncAA掺入。
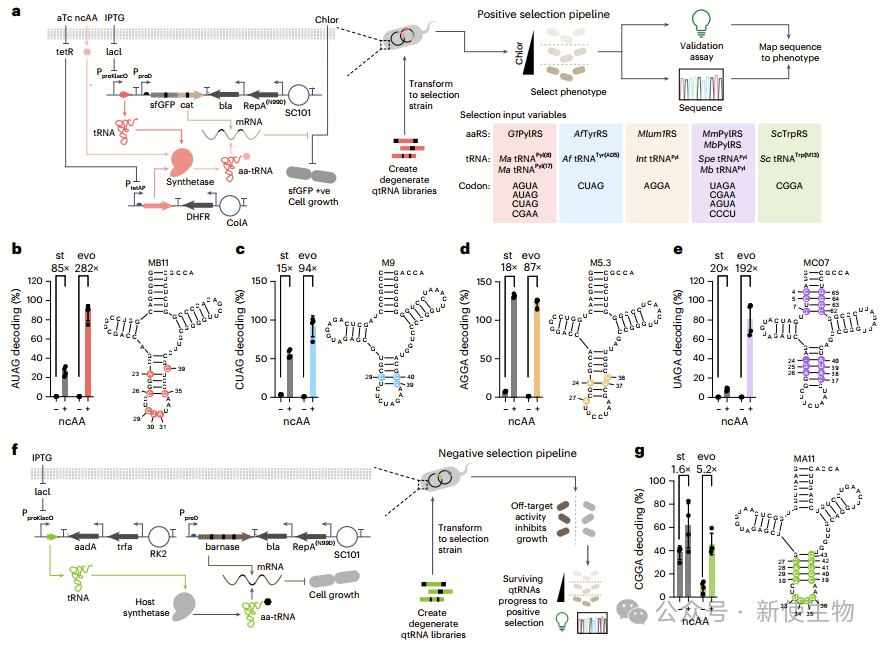
四. 优化合成酶的ncAA识别范围和qtRNA表达是提高四联密码子解码效率的关键步骤
为了实现多重ncAA掺入,作者优化了合成酶的底物范围,以通过少数互不干扰的tRNA-合成酶对引入更多种类的ncAA。通过筛选,他们找到5个优先合成酶的变体,能够有效掺入47种不同的ncAA。这些合成酶可以处理各种取代的苯丙氨酸、酪氨酸、组氨酸衍生物、赖氨酸类似物等。
此外,作者优化了qtRNA的表达,使用多顺反子系统提高了多重四联密码子解码效率,最终实现了在常规大肠杆菌细胞中高效掺入多种ncAA。

五. 具有单一非经典氨基酸的新型大环分子的生物合成
引入非经典氨基酸(ncAAs)到肽类大环分子的策略灵感来源于天然产物的分子及蛋白质功能探针,尽管基因组重写使含有两个ncAAs或羟基酸的大环分子得以生物合成,但这些方法仅限于单一细菌,并且需要细胞内生成硫酯中间体的体外环化。
为解决这些限制,作者采用了一种基于细胞内合成的策略,使用优化的qtRNA-合成酶系统在大环肽中引入多样的ncAAs,并成功生成了多种包含单一ncAA的大环分子,证明了该策略的有效性。

六. 含有多达三种独特非经典氨基酸的新型大环分子
作者优化了四联体密码子解码,实现了含有多达三个不同非经典氨基酸(ncAA)的大环肽的细胞内生物合成。通过共表达多种优选合成酶,成功生成72种独特大环分子,展示了高效的ncAA引入和强大的解码过程性,为开发具有多种化学性质的新型大环肽提供了基础。

总结
本文开发了的策略显著提高了大肠杆菌中四联体密码子的ncAA整合效率。通过这一策略,作者发现了12对正交的tRNA-合成酶组合,并优化了五对以整合多种ncAA。最终,实现了多达三种ncAA的大环肽体内合成。这一方法推动了多重遗传码扩展和新型生物分子的发现。
新使生物NeoRibo推出国内首款超高分辨率翻译组Ribo-seq建库试剂盒。
我们的Ribo-seq核糖体印迹分析技术(QEZ-seq®)适用于哺乳动物、植物、真菌等多类物种,无需复杂操作,建库只需2小时。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
简便快速的操作让翻译组分析不再设有门槛,每位科研学者都能轻松上手。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可联系杭州新使生物有限公司

感谢关注杭州新使生物,polysome profiling


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们