2025年6月12日,在Science期刊上,Weingarten-Gabbay等人报道了一种大规模并行的核糖体印迹分析(ribosome profiling)方法,可以在一次实验中筛查数百种病毒的翻译区域。
点击图片查看详细解读
2025年7月15日,Iris Marchal针对该论文在Nature Biotechnology发表了一篇题为“Profiling translated regions in viral genomes at scale”的点评文章。

点评整理
本文中,研究人员将一个寡核苷酸合成文库转染到两种人类细胞系中,该文库中的每条寡核苷酸包含一个200个核苷酸的病毒序列,两端连接固定引物,并克隆至一个过表达载体中。他们所设计的寡核苷酸涵盖了来自679种病毒基因组的3976个基因的5′ 非翻译区和起始编码区。
通过Ribo-seq核糖体印迹分析,研究者共识别出4208个非经典ORF(non-canonical ORF)。当他们将合成文库中的核糖体足迹分布与四种天然病毒感染中的分布进行比较时,发现两者的足迹位置高度一致。
由于非经典ORF可编码用于人类白细胞抗原I类(HLA-I)呈递的多肽,从而激发强烈的T细胞免疫反应,它们有望成为疫苗研发的新靶点。该方法在巨细胞病毒和痘病毒中识别出了来自七个非经典ORF的HLA-I多肽。
此外,研究人员还发现了数百个上游ORF,这些ORF可能会影响病毒蛋白的翻译起始过程。
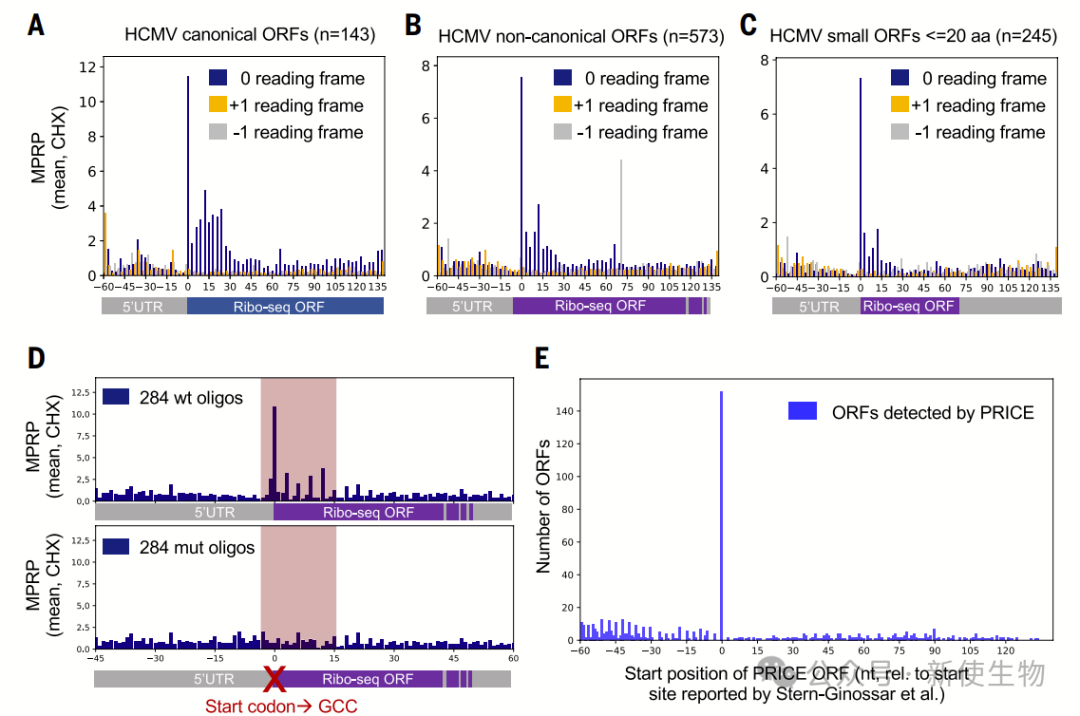
尽管这种方法不能完全模拟病毒自然感染时复杂的细胞环境,但它为快速识别病毒基因组中的翻译区域提供了有力工具,而且无需培养病毒,从而避免了对高等级生物安全实验室的需求,可用于研究高致病性病毒。
我们能够针对微量细胞或组织,如卵母细胞、卵巢、临床穿刺样品等产出高质量翻译组数据结果。
超高的准确性为研究非经典的开放阅读框(ORFs)提供极大便利,提高微肽(肿瘤新生抗原)的挖掘效率。
另外新使生物提供多物种多聚核糖体分析(Polysome profiling),了解更多翻译组技术信息可登录 www.neoribo.com
点击图片查看
点击图片查看


 关于我们
关于我们 产品中心
产品中心 技术服务
技术服务 技术中心
技术中心 联系我们
联系我们